4.4. Visualizaciones#
El análisis visual de los datos es discutiblemente uno de los aspectos más importantes del proceso analítico. En python existen múltiples librerías para generar visualizaciones informativas y hermosas, en esta libreta aprenderás los aspectos básicos de cómo utilizar dos de ellas matplotlib y seaborn.
Trabajaremos con los mismos datos que hemos estado analizando palmer penguins.
4.4.1. Cargar librerías#
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
print('Importaciones listas')
Importaciones listas
4.4.2. Carga de datos#
url = 'https://raw.githubusercontent.com/raphaelvallat/pingouin/refs/heads/main/src/pingouin/datasets/penguins.csv'
df = pd.read_csv(url)
df.head()
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | |
|---|---|---|---|---|---|---|---|
| 0 | Adelie | Biscoe | 37.8 | 18.3 | 174.0 | 3400.0 | female |
| 1 | Adelie | Biscoe | 37.7 | 18.7 | 180.0 | 3600.0 | male |
| 2 | Adelie | Biscoe | 35.9 | 19.2 | 189.0 | 3800.0 | female |
| 3 | Adelie | Biscoe | 38.2 | 18.1 | 185.0 | 3950.0 | male |
| 4 | Adelie | Biscoe | 38.8 | 17.2 | 180.0 | 3800.0 | male |
4.4.3. Análisis con matplotlib#
Retomaremos la última figura del tema de descriptivos básicos. Pudimos usar pandas para separar por color las tres especies pero no sabíamos cuál era cual. Retomaremos esto con matplotlib.
Por favor revisa los acordeone y la documentación de la librería.
Importante
En particular revisa la guía de inicio rápido de matplotlib que puedes encontrar aquí: https://matplotlib.org/stable/users/explain/quick_start.html
Primero, volvamos a hacer la gráfica sin color.
plt.scatter( # matplotlib también tiene una función para la gráfica de dispersión
x=df['bill_length_mm'],
y=df['bill_depth_mm'],
)
<matplotlib.collections.PathCollection at 0x11f880b1d30>
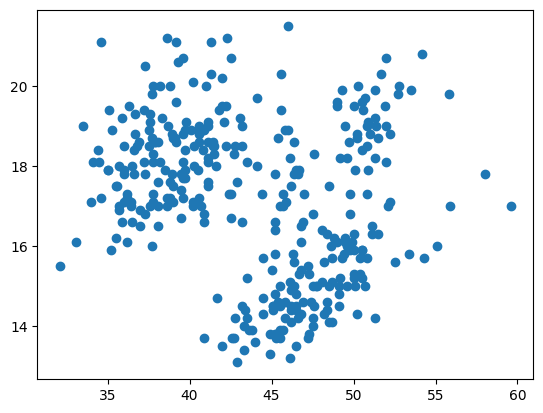
Podemos ver nuevamente los conglomerados, vamos a colorearlos, pero primero veamos el algoritmo de cómo lo vamos a hacer.
flowchart TD;
A[Separar especies] -->
B(Por cada una) -->
C[Generar dispersión] -->
D[Dar color]
Sin embargo, primero entonces necesatamos saber cómo separar a las especies.
En pandas podemos generar un filtro de la siguiente manera:
filtro = df[columna] == valor
filtrados = df[filtro]
Probémoslo seleccionando a los Gentoo.
es_gentoo = df['species'] == 'Gentoo'
gentoo = df[es_gentoo]
gentoo.head()
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | |
|---|---|---|---|---|---|---|---|
| 220 | Gentoo | Biscoe | 46.1 | 13.2 | 211.0 | 4500.0 | female |
| 221 | Gentoo | Biscoe | 50.0 | 16.3 | 230.0 | 5700.0 | male |
| 222 | Gentoo | Biscoe | 48.7 | 14.1 | 210.0 | 4450.0 | female |
| 223 | Gentoo | Biscoe | 50.0 | 15.2 | 218.0 | 5700.0 | male |
| 224 | Gentoo | Biscoe | 47.6 | 14.5 | 215.0 | 5400.0 | male |
Ahora veamos qué pasa si lo graficamos
plt.scatter(
x=gentoo['bill_length_mm'],
y=gentoo['bill_depth_mm'],
)
<matplotlib.collections.PathCollection at 0x11f8a2c6350>
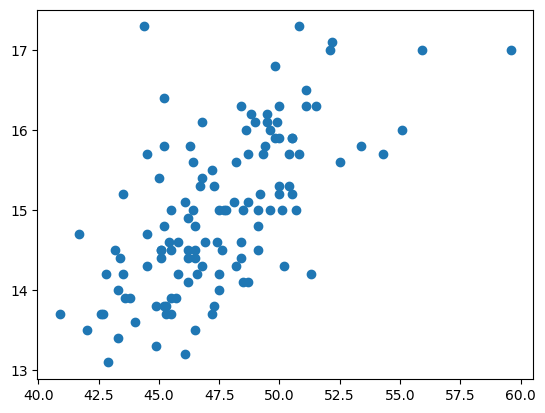
Intenta buscar este patrón en el grafico de todas las especies.
Ahora apliquemos esto en un bucle for.
especies = ['Adelie', 'Gentoo', 'Chinstrap']
for especie in especies:
filtro = df['species'] == especie
datos_especie = df[filtro]
plt.scatter(
x=datos_especie['bill_length_mm'],
y=datos_especie['bill_depth_mm'],
label=especie, # paso clave
)
plt.legend() # le dice a matplotlib que ponga la leyenda.
# mira la magia
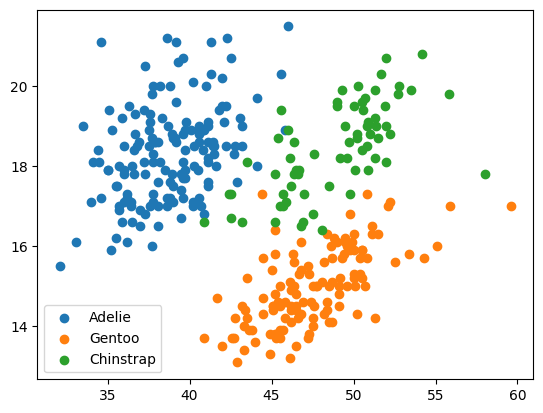
Como ves, a pesar de que generamos tres «gráficos» diferentes, uno por cada especie, en realidad todos se fueron a la misma figura.
4.4.4. Análisis con seaborn#
Ahora, solo para calentar motores, miremos como se ve esto con seaborn.
sns.scatterplot(
data=df, # todos los datos
x='bill_length_mm', # solo el nombre, no los datos
y='bill_depth_mm', # igual
hue='species' # columna para separar colores
)
<Axes: xlabel='bill_length_mm', ylabel='bill_depth_mm'>
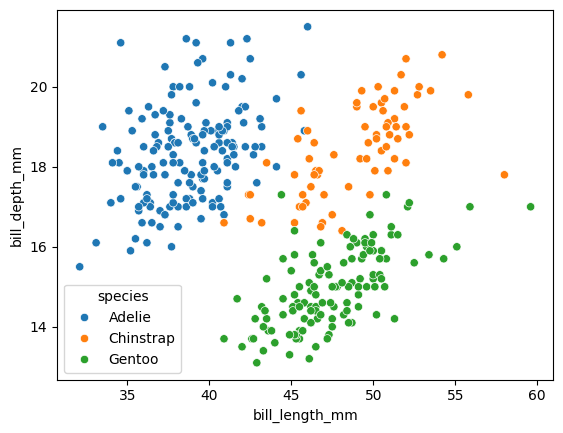
Como ves seaborn abrevia en una sola función de fácil uso, lo que hicimos con plt. Incluso mejor, porque agrega los rótulos de los ejes, cosa que nosotros no hicimos con plt.
4.4.4.1. Las cuatro principales#
Seaborn ofrece tres funciones principales que concentran un enorme porcentaje de nuestras necesidades de visualización:
Función |
Documentación |
Uso |
|---|---|---|
|
Analizar la relación entre variables. |
|
|
Visualizar modelos de regresión lineal (GLM) |
|
|
Visualizar variables categóricas y su relación con cuantitativas |
|
|
Visualizar distribuciones |
A continuación veremos solo algunos ejemplos de cómo utilizarlas.
Por favor revisa la documentación de cada una.
4.4.4.1.1. Relplot#
Relplot ayuda a entender la relación entre varias variables. Es fenomenal para el análisis exploratorio.
sns.relplot(
data=df,
x='body_mass_g',
y='flipper_length_mm',
hue='species',
style='island',
col='sex',
)
<seaborn.axisgrid.FacetGrid at 0x11f880b23c0>
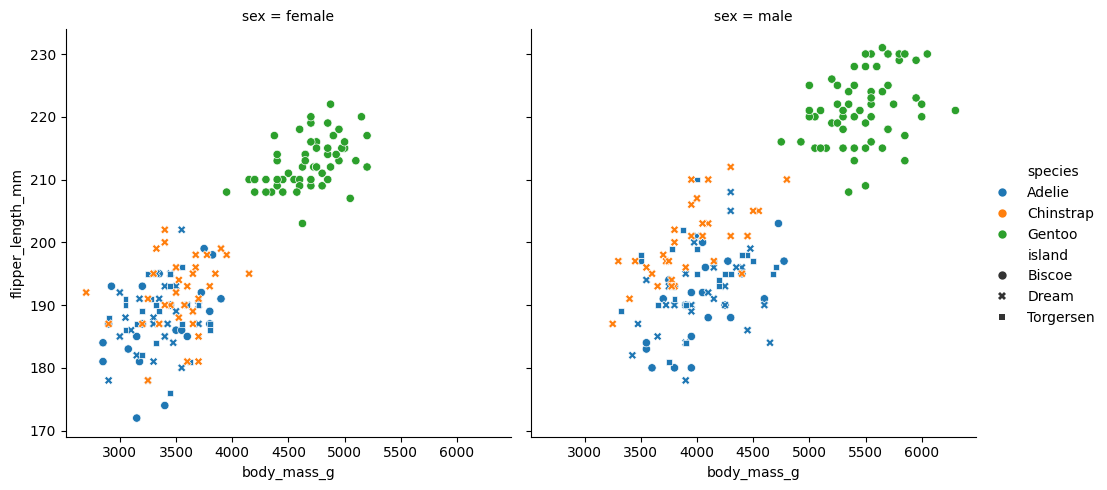
Observa como con una sola función obtenemos un montón de información. Vemos que los Gentoo, son generalmente más grandes y con aletas más grandes. En las tres especies existe dimorfismo sexual, los machos son en general más grandes, pero la diferencia es menos dramática en los Adelie y los Chinstrap. Además, podemos ver que los gentoo solo viven en Biscoe, mientras que los chinstrap solo viven en Dream, sin embargo, los Adelie viven en las tres islas.
4.4.4.1.2. LMplot#
LMplot (linear model plot) ayuda a visualizar modelos regresivos. Por ejemplo, qué pasa si queremos averiguar el tamaño total del pingüino, con base en la medición de su pico, sexo y especie.
\(peso \sim pico + sexo + especie\)
sns.lmplot(
data=df,
x='bill_length_mm',
y='body_mass_g',
hue='species',
col='sex',
)
<seaborn.axisgrid.FacetGrid at 0x11f8b690910>
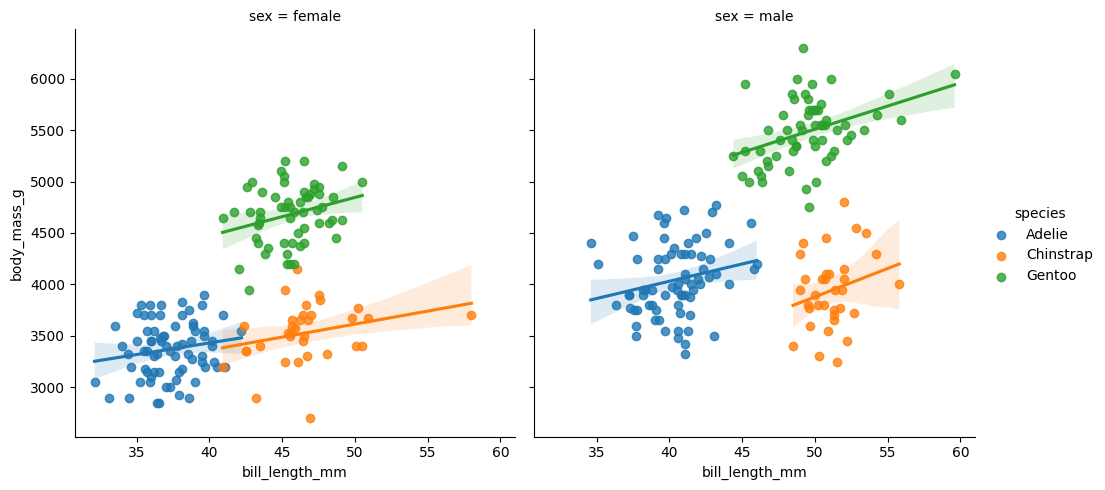
4.4.4.1.3. Catplot#
Catplot nos ayuda a visualizar variables individuales y categorías.
sns.catplot(
data=df,
x='body_mass_g',
y='species',
hue='species'
)
<seaborn.axisgrid.FacetGrid at 0x11f8b730e10>
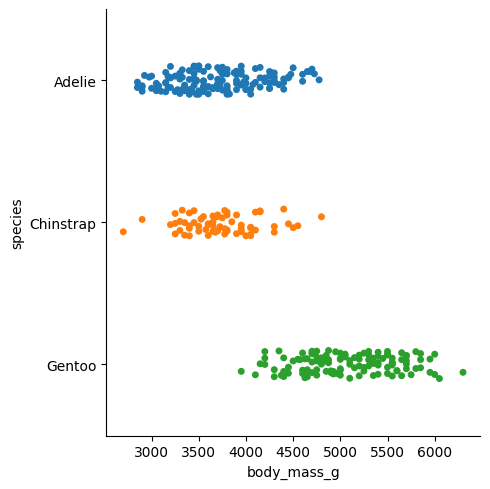
sns.catplot(
data=df,
x='body_mass_g',
y='species',
hue='species',
kind='box',
col='island'
)
<seaborn.axisgrid.FacetGrid at 0x11f8bc1c7d0>
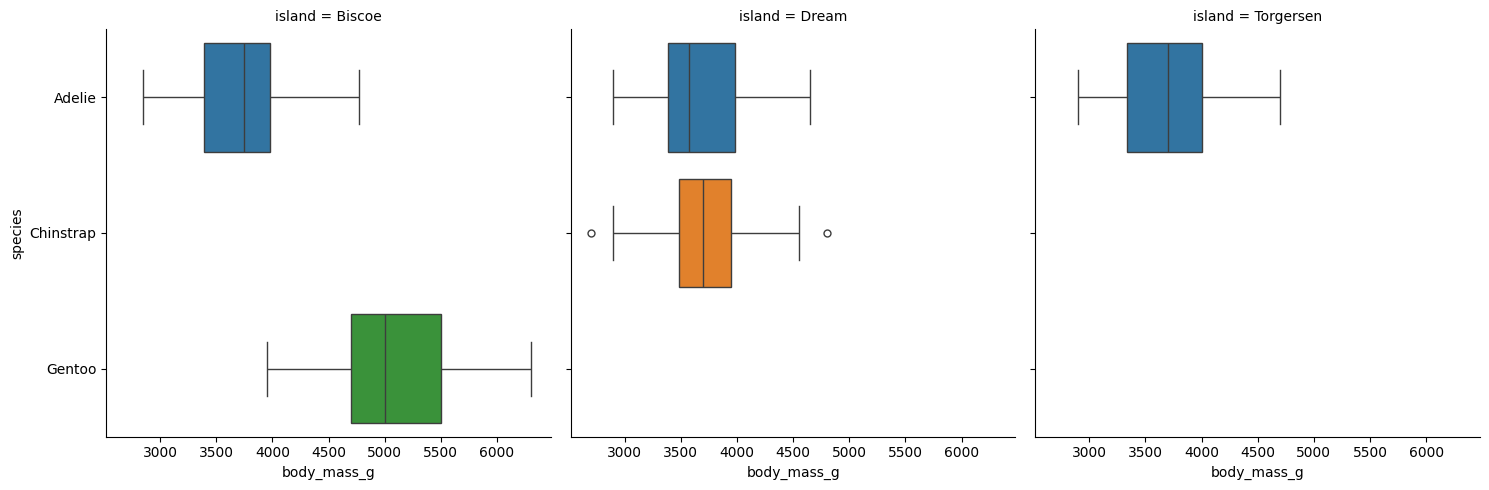
4.4.4.1.4. Displot#
Displot nos ayuda a visualizar distribuciones
sns.displot(
data=df,
x='body_mass_g',
hue='species',
kind='kde',
)
<seaborn.axisgrid.FacetGrid at 0x11f8bd7f250>
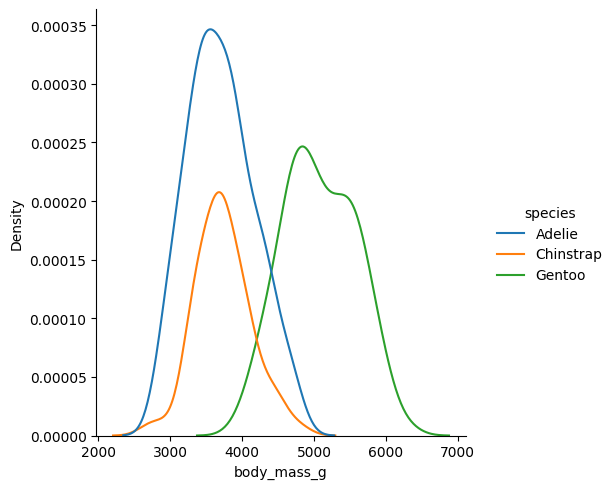
También funciona con distribuciones bivariadas
sns.displot(
data=df,
x='bill_length_mm',
y='bill_depth_mm',
hue='species',
kind='kde',
)
<seaborn.axisgrid.FacetGrid at 0x11f8be15d10>
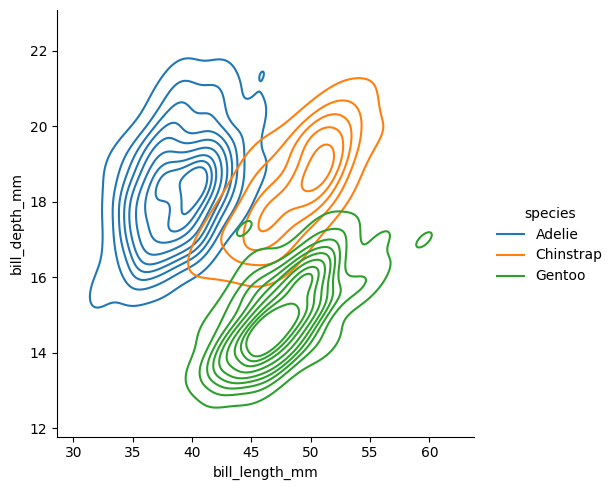
Parece que los Adelíe tienen picos más cortos y anchos, mientras que los Gentoo tienen picos más angostos y largos, aunque los Chintrap se traslapa con las otras dos especies.
4.4.4.1.5. Más sobre seaborn#
Seaborn tiene mucho más que ofrecer, pronto veremos más usos pero vale la pena que conozcas y domines estas cuatro funciones.
4.4.5. Ejercicio#
Revisa la documentación de
seaborn. Particularmente su galería para que veas todo lo que puede hacer.Intenta generar nuevos gráficos para continuar analizando el dataset de palmer penguins.

