6.5. Modelos lineales#
6.5.1. Librerías#
import seaborn as sns
import bambi as bmb
import arviz as az
print('Librerías importadas')
Librerías importadas
6.5.2. Datos#
En esta libreta utilizaremos un modelo lineal múltiple. Utilizaremos un dataset incluido en bambi. Este ejemplo está basado en esta libreta.
Los datos que utilizaremos son de un estudio poblacional en el que se midieron docenas de índices de personalidad en adultos. Este subset contiene cinco predictores numéricos que son sumas de puntuaciones de cinco dimensiones de la personalidad. Las dimenciones son:
O = Apertura a la experiencia
C = Responsabilidad/conciencia
E = Extraversión
A = Amabilidad/Afabilidad
N = Neuroticismo
Puedes leer más de este dataset aquí:
Goldberg, L. R. (2008). The Eugene–Springfield community sample: Information available from the research participants (ORI Technical Report No. 48). http://ipip.ori.org/ORI_TechnicalReport_ESCS_Mar08.pdf
data = bmb.load_data("ESCS")
data.head()
| drugs | n | e | o | a | c | hones | emoti | extra | agree | consc | openn | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1.857143 | 117 | 102 | 122 | 115 | 89 | 3.43750 | 3.46875 | 3.68750 | 3.28125 | 3.18750 | 3.96875 |
| 1 | 3.071429 | 69 | 92 | 142 | 140 | 132 | 4.21875 | 2.59375 | 3.56250 | 3.34375 | 3.84375 | 3.87500 |
| 2 | 1.571429 | 69 | 101 | 117 | 148 | 173 | 3.87500 | 3.46875 | 2.81250 | 2.75000 | 3.84375 | 3.25000 |
| 3 | 2.214286 | 87 | 99 | 130 | 135 | 134 | 3.68750 | 3.09375 | 2.93750 | 3.09375 | 3.62500 | 3.96875 |
| 4 | 1.071429 | 50 | 116 | 100 | 117 | 130 | 4.12500 | 2.59375 | 3.40625 | 3.71875 | 3.84375 | 3.56250 |
Nos enfocaremos solo en las primeras 6 variables, las cinco dimensiones y drugs, nuestra variable de salida, un índice autoreportado del consumo de estupefacientes.
6.5.3. Reconocimiento y exploración de los datos#
data.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 604 entries, 0 to 603
Data columns (total 12 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 drugs 604 non-null float64
1 n 604 non-null int64
2 e 604 non-null int64
3 o 604 non-null int64
4 a 604 non-null int64
5 c 604 non-null int64
6 hones 604 non-null float64
7 emoti 604 non-null float64
8 extra 604 non-null float64
9 agree 604 non-null float64
10 consc 604 non-null float64
11 openn 604 non-null float64
dtypes: float64(7), int64(5)
memory usage: 56.8 KB
data.columns
Index(['drugs', 'n', 'e', 'o', 'a', 'c', 'hones', 'emoti', 'extra', 'agree',
'consc', 'openn'],
dtype='object')
6.5.3.1. Visualizaciones#
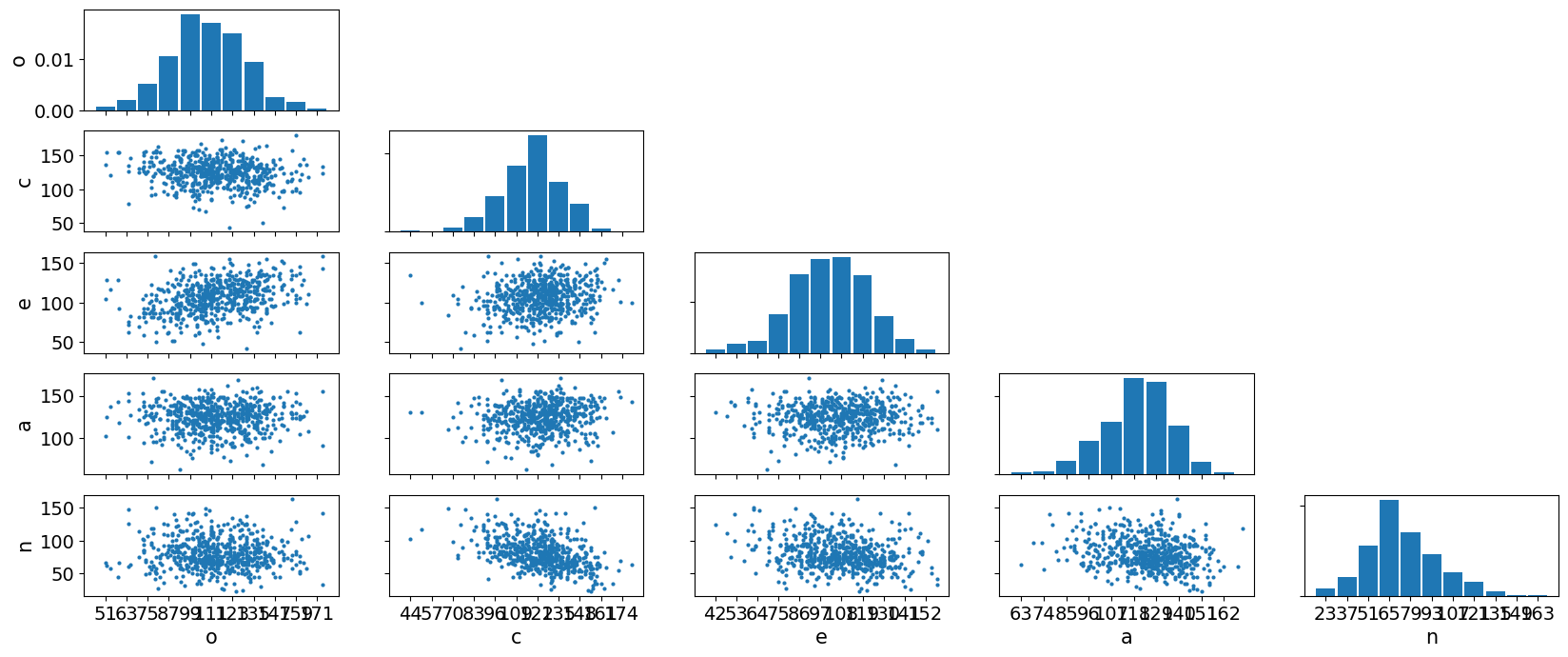
Seaborn tiene una función sns.pairplot que genera una matriz de dispersión con todos los pares, pero dado que estamos utilizando bambi, mostraré cómo utilizar arviz.
az.plot_pair(
data[["o", "c", "e", "a", "n"]].to_dict("list"), # list hace que los valores de cada columna se junten en una lista
marginals=True,
figsize=(20, 8),
);
6.5.4. Modelo#
Como nuestra variable de salida es drugs las dimensiones de la personalidad serán nuestros predictores.
No asumiremos interacción entre ellas ni otros factores externos.
En este ejemplo, dejaremos que bambi seleccione los priors, que serán por defecto, levemente informativos, lo que permite que la inferencia sea dominada por los datos, no por la probabilidad previa.
6.5.4.1. Preparación de los datos#
model = bmb.Model(
'drugs ~ o + c + e + a + n', # la fórmula
data=data, # los datos
dropna=True, # especificamos que queremos quitar los NaN
family='gaussian', # la familia de la variable dependiente
)
model # lo ponemos para ver la especificación.
Formula: drugs ~ o + c + e + a + n
Family: gaussian
Link: mu = identity
Observations: 604
Priors:
target = mu
Common-level effects
Intercept ~ Normal(mu: 2.2101, sigma: 21.1938)
o ~ Normal(mu: 0.0, sigma: 0.0768)
c ~ Normal(mu: 0.0, sigma: 0.0868)
e ~ Normal(mu: 0.0, sigma: 0.0816)
a ~ Normal(mu: 0.0, sigma: 0.0973)
n ~ Normal(mu: 0.0, sigma: 0.0699)
Auxiliary parameters
sigma ~ HalfStudentT(nu: 4.0, sigma: 0.6482)
result = model.fit(chains=4)
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [sigma, Intercept, o, c, e, a, n]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 20 seconds.
6.5.4.2. Diagnóstico de convergencia#
Cuando corras el código en tu libreta deberías ver que no tuvimos divergencias, pero analicemos la traza.
az.plot_trace(result, kind='rank_bars');
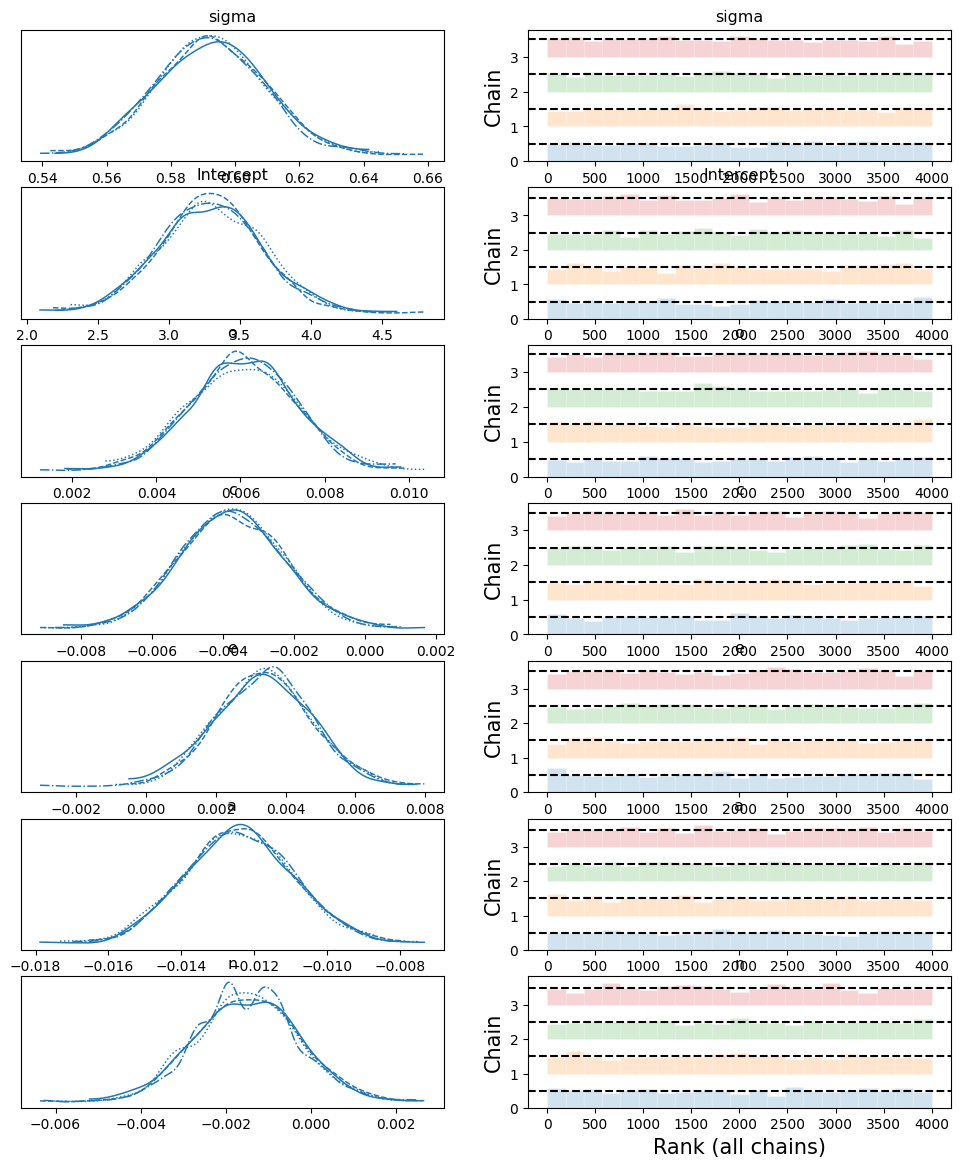
Podemos ver que las cadenas convirgieron exitosamente, lo que indica que las cadenas Markov muestrearon uniformemente todo el espacio de probabilidad posterior.
6.5.4.3. Análisis de coeficientes#
az.summary(result)
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| sigma | 0.593 | 0.017 | 0.561 | 0.623 | 0.000 | 0.000 | 4537.0 | 3350.0 | 1.0 |
| Intercept | 3.292 | 0.361 | 2.646 | 3.990 | 0.005 | 0.006 | 4452.0 | 3126.0 | 1.0 |
| o | 0.006 | 0.001 | 0.004 | 0.008 | 0.000 | 0.000 | 3985.0 | 2885.0 | 1.0 |
| c | -0.004 | 0.001 | -0.007 | -0.001 | 0.000 | 0.000 | 4235.0 | 3138.0 | 1.0 |
| e | 0.003 | 0.001 | 0.001 | 0.006 | 0.000 | 0.000 | 4147.0 | 3016.0 | 1.0 |
| a | -0.012 | 0.001 | -0.015 | -0.010 | 0.000 | 0.000 | 4848.0 | 2964.0 | 1.0 |
| n | -0.001 | 0.001 | -0.004 | 0.001 | 0.000 | 0.000 | 4738.0 | 3467.0 | 1.0 |
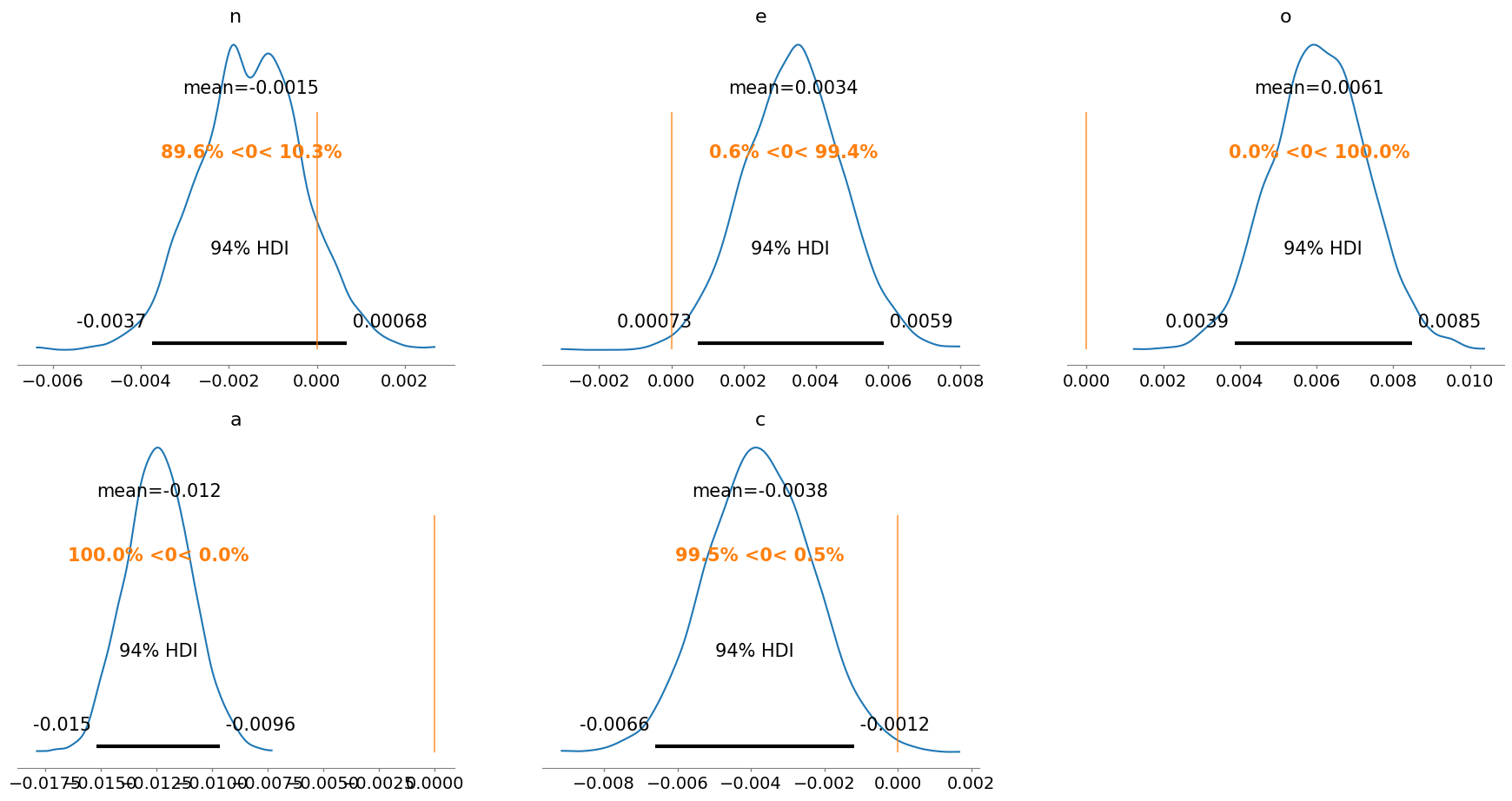
az.plot_posterior(result, ref_val=0, var_names=['n', 'e', 'o', 'a', 'c']);
az.plot_forest(
result,
var_names=['n', 'e', 'o', 'a', 'c'],
combined=True, # combina todas las cadenas
figsize=(8, 3) # mejora el aspecto
);
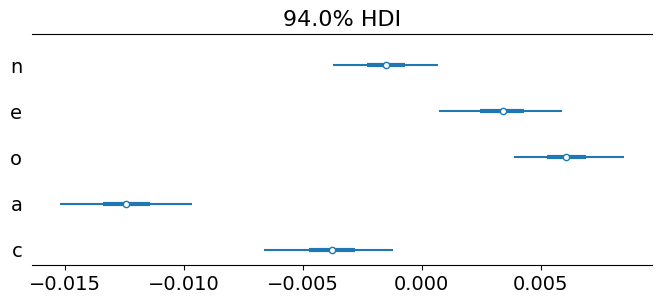
Podemos ver que la dimensión de afabilidad (agreeableness) se correlaciona de forma inversa al consumo de drogas, es un fuerte predictor, de forma similar, la apertura a experiencias (openess) es un predictor positivo.
6.5.5. Conclusión#
Modelar con un marco de trabajo bayesiano es sencillo e intuitivo en python al utilizar librerías de alto nivel como bambi y arviz.
6.5.6. Ejercicios#
Realiza un análisis similar con tus propios datos.
Explora qué otros datasets se incluyen en
bambi.

